Bugün öğrendim ki: DNA analizi, frenginin Avrupa'da değil Amerika'da ortaya çıktığını gösteriyor
Radyoaktif karbon tarihlemesi
Radyoaktif karbon tarihlemesi, Curt-Engelhorn Arkeometri Merkezi'nde (Mannheim, Almanya) gerçekleştirildi. İskelet parçaları ultra filtrelenmiş kolajen kullanılarak işlendi (fraksiyon >30kD)55,56 ve Klaus-Tschira-Arkeometri Zentrum'un MICADAS-AMS cihazı kullanılarak tarihlendirildi. Radyoaktif karbon tarihleri MAMS laboratuvar kodu ile bildirilmiştir.
Kararlı izotop ölçümleri ve deniz rezervuar etkileri
Radyoaktif karbon ve kararlı izotop ölçümleri, Peru'daki Jucusbamba'dan (JUC013), Şili'nin güneyindeki Chonos Takımadaları'ndan (GAP009) ve Arjantin'deki Deán Funes'ten (DFU001) kalıntılar üzerinde yapıldı.
Kalibrasyondan önce, potansiyel bir deniz rezervuar ofseti, tamamen karasal beslenen çağdaş bir bireyden daha yaşlı bir radyokarbon yaşıyla sonuçlanabilecek bireylerden ekstrakte edilen kolajen üzerinde kararlı izotop ölçümleri yapıldı. Veriler, radyokarbon tarihi için kullanılan kolajen özütünden (Mannheim, Almanya, laboratuvar kodu MA) elde edildi ve daha sonra Jena, Almanya'daki Max Planck Jeoantropoloji Enstitüsü'nde (laboratuvar kodu Jena) üretilen aynı kemiğin ikinci bir kolajen özütünün analizi yoluyla doğrulandı. İkinci işlemde, 1 mg kemik kolajeni, kalay kapsüllerde çift olarak tartıldı ve Max Planck Jeoantropoloji Enstitüsü'nde bir Thermo Scientific Flash 2000 Elemental Analizör ile bir Thermo Delta V Advantage Kütle Spektrometresi'ne bağlı olarak yakıldı. İzotopik değerler, daha ağır izotopun daha hafif izotopa oranı (13C/12C veya 15N/14N) olarak, uluslararası standartlara göre binde (‰) δ değerleri olarak rapor edilmiştir (δ13C için Viyana Peedee belemniti (VPDB) ve δ15N için atmosferik N2 (hava)). Sonuçlar, uluslararası standartlara (Uluslararası Atom Enerjisi Ajansı (IAEA)-CH-6: δ13C = −10.80 ± 0.47‰, IAEA-N-2: δ15N = 20.3 ± 0.2‰ ve USGS40: δ13C = −26.38 ± 0.042‰, δ15N = 4.5 ± 0.1‰) ve bir laboratuvar standardına (balık jelatini: δ13C ≈ −15.1‰, δ15N ≈ 14.3‰) göre kalibre edildi. Tekrarlanan analizlere dayalı olarak, bir yıl boyunca uzun vadeli makine hatası δ13C için ±0.2‰ ve δ15N için ± 0.2‰'dir. Genel ölçüm hassasiyeti, balık jelatininin tekrarlarının ölçümü yoluyla incelendi (n = 80, δ13C için ±0.2‰ ve δ15N için ± 0.2‰). İzotopik veriler Ek Tablo 2'de bildirilmiştir. Tüm örnekler, 2.9–3.6 arasında bir C/N oranı, %15–48 oranında %C ve %5–17 oranında %N ile kalite kontrol kriterlerini karşılamıştır57,58.
Jucusbamba'dan (JUC013) bireyin, tamamen karasal bazlı bir C4 diyeti gösteren kararlı izotopları vardı, bu nedenle deniz ofseti dikkate alınmadı. Buna karşılık, Chonos Takımadaları'ndan (GAP009) bireyin, hem C4 hem de deniz diyet girdileriyle uyumlu bir δ13C değeri (−10.5‰) vardı, ancak diyetinde yüksek bir deniz bileşenini işaret eden bir δ15N değeri (+18.2‰). Şili, C3 bitkileriyle hakimdir59, bu nedenle diyetindeki yüksek deniz proteini girişi, kararlı izotoplardaki kaymaya muhtemelen katkıda bulunmuştur. İlginç bir şekilde, bu birey için δ13C değeri, bölgeden yüksek trofik seviyeli deniz etoburlarından, örneğin Yesner vd.60 tarafından özetlenen Tierra del Fuego'dan deniz aslanı verilerinden daha fazla zenginleşme göstermiştir. Deán Funes'ten (DFU001) birey için, yalnızca AMS üzerinde ölçülen δ13C mevcuttu, bu da diyet düzeltmesi için gereken hassasiyetten yoksundur. Buna rağmen, bu saha kıyıdan 600 km'den fazla uzaklıkta yer almaktadır, bu nedenle bir deniz diyet bileşeni oldukça düşük olasılıklı kabul edilmektedir.
Birey GAP009'daki ilişkili radyokarbon ofsetini hesaba katmak için bir modelleme yaklaşımı gerekiyordu. Bu bireyi yorumlamak için doğrudan ilgili izotop verilerinin (örneğin, aynı sahadan faunal örnekler üzerinde yapılan ölçümler) olmaması nedeniyle, beklenen δ13C ve δ15N değerlerinin hem tamamen karasal hem de deniz diyetleri için aralığını yaklaşık olarak belirlemek için bir literatür taraması yapıldı. Karasal bir diyet için δ13C aralığı −22,5 ile −19,5‰ ve deniz diyetleri için −16 ile −12‰ olarak tahmin edilirken, δ15N sırasıyla +7–11‰ ve +14–22‰ olarak belirlendi. Bu aralıklar, Tierra del Fuego için arkeolojik faunal verilerinden tahmin edilmiş ve Yesner vd.60 ve Şili'nin kuzeybatı Patagonya'sından at (Equus andium) ve lama (Hemiauchenia paradoxa) geç Pleistosen kararlı izotop verilerinden özetlenmiştir61. Tarih düzeltmesi, δ13C uç üyeleri −20 ve −12‰ (sırasıyla %100 Karasal ve Deniz) ve δ13N uç üyeleri +10 ve +22‰ (sırasıyla %100 karasal ve deniz) olmak üzere doğrusal enterpolasyon yöntemine göre yapılmıştır62,63. Hem izotoplar kullanılarak enterpolasyon yapılması, tarihlendirmenin bu düzeltmeye genel duyarlılığının kontrol edilmesini sağlamıştır. Bu özellikle önemlidir çünkü: (1) zamansal ve mekansal olarak ilgili biyolojik olarak kullanılabilir bir temel oluşturmak için ilişkili faunal materyal üzerinde izotop verisi yoktu; ve (2) δ13C değerleri, tamamen deniz proteininden oluşan bir diyet için beklenenden daha zenginleştirilmiştir. Modellemenin sonucu, sırasıyla δ13C ve δ15N değerleri kullanıldığında %100 veya %85 deniz diyetini tahmin etmektedir.
Üç bireyin (GAP009, JUC013 ve DFU001) tamamı Güney Yarımküre'de yaşadığı için, radyokarbon sonuçlarını OxCal v4.4.51 kullanarak bir takvim ölçeğine kalibre etmek için Hogg vd.52'nin SHCal20 kalibrasyon eğrisi kullanılmıştır.
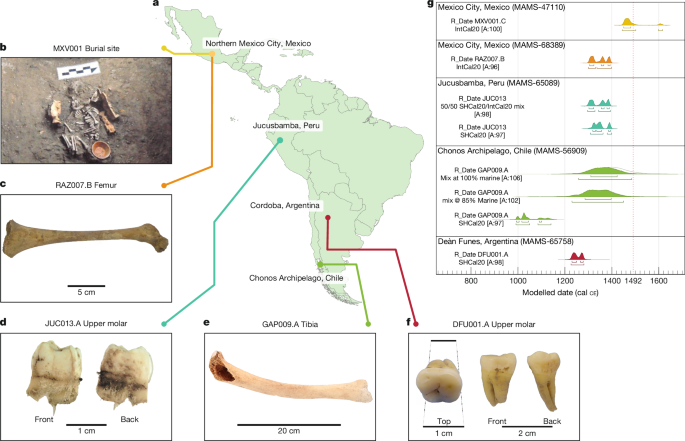
Peru'daki Jucusbamba, Marsh vd.64 tarafından gösterilen bir karışım bölgesi olan Intertropikal Yakınsama Bölgesi'nin (ITCZ) hemen batısında yer almaktadır. Bu nedenle, Kuzey Yarımküre IntCal2053 ve SHCal20 arasında 50/50 bir karışımla, %25'lik bir 1σ hatasıyla, karışık bir kalibrasyonun nispeten belirsiz bir yaklaşımı olarak, etki değerlendirilebilmesi için ikinci bir kalibrasyon yapılmıştır.
ITCZ karışımı için modelleme yapılmadan, kalibre edilmiş tarih, %73,7 olasılıkla 1308–1363 MS ve %21,7 olasılıkla 1380–1399 MS aralıklarına sahip bimodaldir. Modelleştirilmiş kalibrasyon da bimodaldir ve biraz daha erkendir, %40,0 olasılıkla 1296–1328 MS ve %55,5 olasılıkla 1342–1394 MS aralıklarına sahiptir.
Şili'deki Chonos Takımadaları'ndan birey için, iki kalibrasyon SHCal20 ve Marine20 kalibrasyon eğrilerini sırasıyla %85 ± %10 ve %100 ± %10 oranında karıştırdı. Marine20 ayrıca, Calib.org Marine20 veritabanındaki (http://calib.org/marine/) en yakın sekiz noktanın ağırlıklı ortalaması olan −93 ± 30 yıllık yerel bir ΔR düzeltmesine sahipti. %85 modelli düzeltme, %95,4 olasılıkla 1226–1451 MS'ye kalibre edilirken, %100 modelli düzeltme %95,4 olasılıkla 1255–1483 MS'ye kalibre edilmektedir.
Son olarak, Deán Funes, Marsh vd.64 tarafından SHCal20 kullanılarak kalibrasyon gerektiren olarak belirlenen alanın sınırları içinde yer almaktadır. Bu nedenle, modelleme veya başka bir düzeltme gerekmemiştir ve bu nedenle kalibrasyon için yalnızca SHCal20 kullanılmıştır. Bu birey, %95,4 olasılıkla 1226–1281 MS yılları arasında ölmüştür.
Yukarıdakiler için ek sayısal veriler Ek Tablo 2'de bulunabilir. Modelleştirilmiş tarihler Şekil 1g'de gösterilmiştir.
DNA ekstraksiyonu, kütüphane hazırlama ve dizileme
Arkeolojik dokuların işlenmesi, Max Planck Evrimsel Antropoloji Enstitüsü'nün eski DNA temiz odası tesisinde gerçekleştirildi. Kemikten öğütülmüş malzeme, düşük rotasyon ve yüksek torkta bir diş matkabı ile üretildi. JUC013'ün olgunlaşmamış azı dişi için, her biri ekstraksiyon için yaklaşık 50 mg dentin için iki ayrı örnek oluşturmak üzere, kaynamamış köklerden taç girişine doğru toz matkapla delinmiştir. Arjantin ve Şili dokularının alt örneklemesi yerinde (D.A.R., R.B. ve T.v.H. tarafından) yapıldı ve malzeme daha sonra temiz oda tesisinde işlendi. Şili'den biyolojik malzeme koleksiyonu, doğrudan bir protein kinaz sindirim tamponunda çözünen yumuşak dokuları içeriyordu. Özellikle birey GAP009 için, hem subperiostal lezyonun sahasında hem de lezyona bitişik bir konumda toz elde edildi. Birey MXV001, treponemal enfeksiyonla uyumlu patoloji gösteren konumlardaki birden fazla iskelet unsurundan örneklendi. Birey RAZ007, lezyonların gösterildiği oksipital kemikten ve sol femur distal diafizinden örneklendi. Son olarak, birey DFU001, spesifik olmayan enfeksiyonlara bağlanabilen lezyonların gözlendiği sol iliak krestten örneklendi. DNA, kısa DNA parçalarının geri kazanımı için optimize edilmiş silika bazlı bir yöntemle ekstrakte edildi65. Kısaca, 2,0 ml'lik Eppendorf Lo-Bind tüplerde65,66 örnek malzemeye 1–1,8 ml ekstraksiyon tamponu (0,45 M EDTA, pH 8,0, 0,25 mg ml−1 protein kinaz, %0,05 Tween-20) eklenerek ve tüpleri yaklaşık 16 saat 37 °C'de döndürülerek lizatlar hazırlandı. GAP009, DFU001 ve RAZ007 için bu lizat doğrudan kütüphane hazırlama için kullanıldı. MXV001 tozları ve JUC013 için, lizat daha fazla saflaştırıldı ve bir silika membran üzerinden konsantre edildi65. JUC013'ten 10 µl özüt28 kullanılarak çift sarmallı DNA kütüphanesi manuel olarak oluşturuldu, bu daha sonra shotgun tarama kütüphanesi JUC013.A0101'i üretti. Kısmi UDG işlemi67 içeren kütüphaneler, 30 µl özüt kullanılarak MXV001 DNA izolatı için üretildi. Hem GAP009 sindirimlerinden de 150 µl'lük alikotların lizatları, Rohland vd.'de açıklandığı gibi66 silika kaplı manyetik boncuklar ve bağlama tamponu D ile otomatik sıvı taşıma sistemi (Bravo NGS Workstation B, Agilent Technologies) kullanılarak saflaştırıldı. Elusiyon hacmi 30 µl idi. RAZ007, DFU001 ve GAP009 lizatının lezyondan (GAP009.A0101) ve bitişiğindeki bölgeden (GAP009.A0201) tek sarmallı kütüphaneleri, ayrıca JUC013 özütünden 30 µl'den sonraki kütüphaneler (JUC013.0102) ve ek toz (JUC013.A0201; GAP009 için yukarıda açıklandığı gibi ekstrakte edildi) otomasyon yoluyla üretildi. Gansauge vd.'de ayrıntılı olarak açıklanan tek sarmallı DNA kütüphane hazırlamasının29 otomatik bir versiyonu kullanılarak 30 µl özütten DNA kütüphaneleri hazırlandı. Moleküllerin iç kısmındaki urasillleri (kısmen veya tamamen) çıkarmak için kütüphane hazırlama sırasında (fosfatazla birlikte) Escherichia coli UDG ve E. coli endonükleaz VIII eklendi. GAP009.A0301, GAP009.A0401, GAP009.A0501, JUC013.A0102 ve JUC013.A0201 kütüphanelerinin oluşturulması için uygulandı. Tüm ekstraksiyonlar ve kütüphaneler için paralel olarak negatif kontroller üretildi. Kütüphane verimleri ve kütüphane hazırlamanın verimliliği, iki kantitatif PCR analizi kullanılarak belirlendi29.
T. pallidum DNA'sı için hesaplamalı tarama
GAP009 (iki kütüphane), MXV001 (üç kütüphane), DFU001 (bir kütüphane) ve RAZ007 (iki kütüphane) verileri, tüm 'tam' veya 'kromozom' seviyesindeki bakteri genomlarından (3 Kasım 2017'de), 'tam' viral genomlardan (adlarında 'diğer' veya 'bilinmeyen' olmadan) (30 Ekim 2017'de) ve NCBI assembly portalından elde edilen seçilmiş ökaryotik genomlardan oluşan bir MALT69 veritabanı kullanılarak HOPS30 aracılığıyla tarandı. Okuma işlemi, insan okumalarının çıkarılması ve MALT çıktısının değerlendirilmesi için HOPS analizi, nextflow (nf)-core/eager v2.4.432 boru hattı aracılığıyla gerçekleştirildi, nextflow v21.04.170 iş akışı dili üzerine kuruldu. nf-core/eager, dizileme verilerinin kalite kontrolü ve değerlendirmesi için, eşleme verilerini oluşturmanın yanı sıra analiz eden ve SNP çağrıları üreten tekrarlanabilir bir boru hattıdır. nf-core/eager boru hattı, eski DNA için optimize edilmiştir ve adaptörlerin çıkarılması ve okumaların 3′ ucundaki düşük güvenilirlikli baz çağrılarının kırpılması için AdapterRemoval v27171, eşleme için bwa v0.7.1772, düşük eşleme kalitesine sahip okumaların çıkarılması için samtools c1.1273 (<37), çoğaltmaların çıkarılması için dedup v0.21.8 ve okumalardaki hasar yüzdelerini analiz etmek için DamageProfiler74 dahil olmak üzere birkaç altın standart yayınlanmış yazılım paketi kullanır. Bu nf-core/eager boru hattı tarafından kullanılan yazılımların tam listesi Ek Tablo 3'te bulunabilir. Adaylar, HOPS aday profillerine göre değerlendirildi (Ek Şekil 10). JUC013, yukarıda açıklanan yöntemin alternatif bir hesaplamalı yaklaşımı aracılığıyla T. pallidum adayı olarak tanımlandı (Ek Bilgiler, bölüm 3). MALT'ta Treponema cinsine atanan RAZ007'den okumaların sayısı (femur için n = 3 ve oksipital için n = 2), HOPS'ta bir değerlendirmeye izin verecek kadar düşük idi.
T. pallidum DNA'sı için zenginleştirme ve dizileme
Örnek ve kontrol kütüphaneleri, Bravo NGS iş istasyonunda otomatikleştirilmiş iki ardışık çözelti içi yakalama75 turunda TPA DNA'sı için zenginleştirildi. Hem zenginleştirilmemiş hem de zenginleştirilmiş kütüphane ürünleri, tek uçlu 75 bp kimyası kullanılarak bir Illumina HiSeq 4000 üzerinde dizilenmiştir.
T. pallidum yakalama verilerinin analizleri
Ek Bilgiler, bölümler 6–8'e bakın.
İnsan ata analizi
Shotgun ve insan zenginleştirilmiş yakalama verilerini nf-core/eager v. 2.3.432 kullanarak analiz ettik. Adaptör dizilerinin kırpılması ve adaptör dimerlerinin ve düşük kaliteli dizileme okumalarının çıkarılması için AdapterRemoval v.271 kullanıldı (min uzunluk = 30; min baz kalitesi = 20). Ön işlenmiş diziler, Genom Referans Konsorsiyumu76'dan BWA v. 0.7.1272 ve 32'lik bir çekirdek uzunluğu kullanılarak insan genomu montajı GRCh37 (hg19)'a eşlendi. Eski DNA parçalarının özgünlüğünü değerlendirmek için mapDamage 2.077 kullanılarak eski DNA'nın tipik C'den T'ye yanlış dahil etme frekansları elde edildi. Analiz edilen bireylerin genetik cinsiyeti, hedeflenen SNP'lerdeki kromozom uzunluğuna göre normalize edilen ortalama X kromozomal ve Y kromozomal kapsamın ortalama otozomal kaplamaya oranı hesaplanarak SNP yakalama verileri kullanılarak atandı78. 0,35 ile 0,55 arasında bir X oranına ve 0,4 ile 0,7 arasında bir Y oranına sahip örnekler erkek olarak doğrulandı (JUC013, DFU001, MXV001 ve RAZ007). Erkeklerin her X kromozom pozisyonunda homozigot olması beklendiği için79, nükleer kontaminasyonu tahmin etmek için ANGSD v. 0.935 (nf-core/eager'da uygulandığı gibi) kullanıldı. Kontaminasyon tahminleri, analiz edilen tüm bireyler için 0,0 ± 0,0 değerini verdi. Her bireyin birleştirilmiş dizileme verilerinden bir yığılma dosyası oluşturmak için samtools mpileup (parametreler –q 30 –Q 30 –B) kullandık ve bireyleri genotiplemek için özel bir komut dosyası (pileupCaller v. 8.2.280) kullandık, psödo-haploit rastgele çekiliş yaklaşımı kullanarak. Daha fazla popülasyon genetiği analizi için yalnızca >10.000 SNP çağrısı yapılan bireyler tutuldu (GAP009: 156.533; JUC013: 761.151; MXV001: 1.027.622; DFU001: 43.818 (İnsan Kökenleri panelinde)), (daha düşük sayıda çağrılan SNP nedeniyle RAZ007 hariç tutuldu, 2.134).
Bireylerin mitokondriyal genomları için konsensus dizileri, shotgun ve post-yakalama zenginleştirmeden (sadece MXV001 için post-yakalama zenginleştirme) elde edilen dizileme okumalarının revize edilmiş Cambridge referans dizisine81 eşlenmesi yoluyla belirlendi. Mitokondriyal DNA (mtDNA) dizileri hizalandı ve Geneious Prime 2021.1.1 (https://www.geneious.com) ile manuel olarak incelendi. Ortaya çıkan diziler için, 30'un üzerinde olasılığa sahip pozisyonları filtreledik ve karşılık gelen mtDNA haplo gruplarını atamak ve doğrulamak için HaploGrep282 ve HAPLOFIND83 kullandık. Hizalanmış mtDNA genomları, literatürden elde edilen diğer mevcut genomlarla karşılaştırıldı. Bu sefer mtDNA bilgileri ve Mitoverse84'ten Haplocheck–Kontaminasyon Tespiti v1.0.0 kullanılarak insan verilerinin işlenmesinden sonra yeni bir kontaminasyon tahmini yapıldı. Kontaminasyon durumu ve kontaminasyon seviyesinde beş birey için elde edilen sonuçlar, RAZ007 hariç algılanamamaktadır (mtDNA referansındaki ortalama kapsam: 549X (MXV001), 1252X (DFU001), 721X (GAP009), 53X (JUC013) ve 224 (RAZ007, kontaminasyon seviyesi %4,5)). Y-haplogrubu atamaları için, ISOGG Y-DNA Haplogrubu Ağacı'nda (v15.73; https://isogg.org/tree/) listelendiği gibi Y-kromozom SNP'lerine eşlenen her birey için okumaların yığılmalarını oluşturduk. Daha sonra, Rohrlach vd.'de açıklandığı gibi85, Y-kromozom haplo gruplarının filogenisini boyunca yukarı akış mutasyonlarının varlığını değerlendirdikten sonra alınan en aşağı akış SNP'sine dayanarak her birey için Y-kromozom haplo gruplarını manuel olarak atadık.
JUC013, MXV001, DFU001 ve GAP009 için dizileme verilerini, Ek Tablo 27'de özetlenen yayınlanmış genomik veri kaynaklarıyla birleştirdik. Avrupa/Afrika ata bileşenleri ile karışımı araştırmak için (Şekil 4a), 7271 eski ve modern bireyden (593.115 SNP) oluşan bu dünya çapındaki referans setinden bir ana bileşen analizi (PCA) oluşturuldu. Düşük kapsamı nedeniyle (1240 K referans panelinde 10.000'den az çözülmüş SNP) bu analizden RAZ007 hariç tutuldu.
Beş kıta bölgesinden atalardan kalma popülasyon kümesini, temas sonrası dönemde doğmuş olmanın açık bir göstergesi olacak olmayan Yerli Amerikalı popülasyonlarla karışımı test etmek için karışım oranlarını tahmin etmek için kullandık: Örneklerimize Yerli Amerikalı katkısını değerlendirmek için genetik havuzunun bir parçası olarak Huichol43, Maya, Karitiana86 ve Mixtec87'yi dahil ettik; Afrika bileşeni, Yoruba88, Esan, Mende ve Luo78 gruplarından bireylerin genetik verilerini kullanarak tahmin edildi; İspanyol87,89 ve Fransız86,89, Avrupa genetik katkısını modellemek için kullanıldı; Doğu Asya genetik katkısı Han ve Kamboçyalılardan88 oluşuyordu ve yakın Okyanusya bileşeni Papualılardan88 alınan genotiplerle tahmin edildi. Daha sonra, modelimiz için en iyi K'yi (yani, en düşük çapraz doğrulama hatasına (CVE) sahip olan; K = 5) ve modelleme yapılan her bir ebeveyn popülasyonunda en üst düzeye çıkarılan bileşenlerin karışım oranlarını hesaplamak için ADMIXTURE90 ve AdmixturePlotter91 kullandık (Şekil 4d,e). Analiz edilen bireylerde Yerli Amerikalı ata soyunun görselleştirilmesini daha da geliştirmek için, yalnızca Amerika'nın Yerli popülasyonlarını atalardan kalma popülasyonlar olarak kullanarak başka bir ADMIXTURE çalışması yapıldı: Arktik, Kuzey Amerika, Karayipler, Mezoamerika ve Güney Amerika bölgelerinden popülasyonlar atalardan kalma olarak dahil edildi (Şekil 4f,g). En düşük CVE, K = 5'te tahmin edildi. Sonuçlar, analiz edilen bireyler için bağlamsal bilgilerin sağladığı coğrafi bölgeler ve arkeolojik dönemler için beklenen karışım oranlarını verdi.
ADMIXTURE analizinde önerilen genetik ilişkileri ve karışım olaylarını değerlendirmek için, Poseidon çerçevesinden Xerxes CLI yazılımını kullanarak F-istatistik analizleri gerçekleştirdik. Afrikalı bir dış grup türünden ayrıldıklarından sonra Yerli Amerikalı test popülasyonları ve analiz edilen her birey arasındaki paylaşılan genetik sürüklenme miktarını ölçmek için F3(dış grup; test, X) biçiminde F3 testleri gerçekleştirdik (Şekil 4a–c), burada X analiz edilen her bireyi, test analizlerimize dahil edilen Yerli Amerikalı popülasyonlarının her birini ve dış grup Kongo88'den Mbuti bireylerini ifade etmektedir.
Virülans faktörü analizi
Eski ve modern treponemal genomlar arasında olası fonksiyonel farklılıkları çıkarmak için, pallidum (sifiliz) ve pertenue (yaws)/endemicum (bejel) soy hatlarında ve dış grup T. paraluiscuniculi93,94,95'te (Ek Tablo 25) virülans profilleriyle potansiyel olarak ilişkili genlerin varlığını veya yokluğunu araştırdık. Bunun için, SAMtools73 ile bir eşleme kalitesi filtresinin uygulanmasından önceki BAM dosyaları, daha önce yapıldığı gibi26,96, Nichols referans genomunda (NC_021490.2) (Ek Tablo 26) açıklanan 68 kromozomal gen boyunca kapsamı hesaplamak için kullanıldı. Her araştırılan gen boyunca kapsam oranları BEDtools97 ile hesaplandı ve daha sonra R sürüm 4.2.2'nin ggplot2'sini kullanarak bir ısı haritasına çizildi (Genişletilmiş Veri Şekil 3 ve Ek Tablo 26). Ayrıca, çağrılan SNP'lerin (SNP çağırma stratejisi hakkında ayrıntılar için Ek Bilgiler, bölüm 7.3'e bakın) olası fonksiyonel etkisini, sekiz eski ve 53 modern treponemal genom boyunca çıkarmak için snpEff sürüm 3.1i [build 2012-12-12]98 programını kullandık. Çıkarılan SNP etkileri, analizlerimiz için oluşturulan tüm treponemal genomların (n = 61) karşılaştırmalı SNP tablosuna MultiVCFAnalyzer v0.85.299 kullanılarak entegre edildi. Tablo, eski genomlar MXV001 (Ek Tablo 21), DFU001 (Ek Tablo 22) ve JUC013/GAP009 (Ek Tablo 23) ortaya çıkan üç yeni rapor edilen soy hatlarında bulunan SNP'leri ve etkilerini ve tüm modern TPA (sifiliz) çeşitliliğinde türetilenleri (Ek Tablo 24) tanımlamak için filtrelendi.
Moleküler tarihleme
Bir moleküler tarihleme analizinin potansiyelini araştırmak için, veri setimizde zamansal sinyalin varlığını iki farklı yaklaşım kullanarak resmi olarak değerlendirdik. İlk olarak, Clockor239 ile uygulanan kök-uç regresyonu gerçekleştirdik (Ek Şekil 18). Bu, kök ile genetik mesafe arasında (R2 = 0,25) pozitif bir korelasyon gösterdi. TPA ve TPE/TEN kladları için bağımsız olarak uygulanan regresyonlar, her iki soy hattında da benzer evrimsel oranlar önerdi (sırasıyla 6,7 × 10−8 ve 8,4 × 10−8 ikame/site/yıl; Ek Şekil 18), ancak yerel saat modeli küresel saat modeline göre biraz daha tercih edildi (Bayes bilgi kriteri (BIC) = sırasıyla −1.250 ve −1.254). İkinci bir adımda, yol örneklemesi41 kullanılarak, BEAST 2 model seçimi paketinde38 uygulanan şekilde katı bir saat ve ilişkisiz log-normal gevşemiş bir saati karşılaştırdık. Bu durumda, model karşılaştırma için skyline birleşik dağılımındaki bilinen sorunlar nedeniyle100, üstel bir birleşik dağılım ağacı önseli kullanıldı. Son popülasyon boyutu için 0 ile 1 milyon arasında uygun bir düzgün önsel kullanıldı. Bu analizler için, eski genomların tarihleri ortanca tahminlerine sabitlendi. Her modelin marjinal olasılığı daha sonra yol örneklemesi40 kullanılarak tahmin edildi. Başlangıçta her biri 3 milyon yineleme olmak üzere ayarlanan (50% yakma süresi dahil) 10 milyon yineleme ön yakma süresi ve 100 adım kullandık, bu daha sonra sonuçta ortaya çıkan Bayes faktör tahmini yakınsadığından emin olmak için aynı uzunlukta uzatıldı. İkincisi, gevşemiş saat modeli için ezici bir destek gösterdi (log BF = 167). Son olarak, zamansal sinyalin Bayes değerlendirmesini (BETS)40 gerçekleştirdik. Daha önce seçilen gevşemiş saat modeli, daha sonra tüm örneklerin 0 bp olarak ayarlandığı ve saat oranının bir olarak sabitlendiği, diğer tüm parametreler aksi takdirde aynı olan bir izokron modelle karşılaştırıldı. Bunun için, yukarıda açıklandığı gibi yol örneklemesi40 kullanıldı. Sonuçlar, veri setimizde önemli bir zamansal sinyalin varlığını doğrulayan, heterokronik model için güçlü bir destek gösterdi (log BF = 11,1).
Veri setimizde önemli bir zamansal sinyalin değerlendirilmesinin ardından, yukarıda açıklanan 2104 SNP'yi içeren hizalamaya dayalı olarak BEAST 2.6.738 kullanılarak zaman kalibre edilmiş bir filogenetik ağaç tahmin edildi (Şekil 3 ve Ek Tablo 20). Tüm filtreleme adımları uygulandıktan sonra kalan sabit site sayısını hesaba katmak için filtrelenmiş bir hizalama kullanıldı. Ağaç, örnek tarihleme bilgileri kullanılarak kalibre edildi. Modern suşlar için izolasyon tarihleri kullanıldı. Eski suşlar için, tarih bilgisi, diyet modelli 2σ 14C tarih aralıklarına veya tarihsel verilere (Ek Tablo 28) dayalı olarak sınırlandırılmış düzgün önsel dağılımlar olarak girildi. Dört ayrı kategori101 kullanarak gama dağıtılmış bir hız heterojenliğine sahip genel zaman tersine çevrilebilir (GTR) ikame modeli, veri setindeki her konum için %25'lik eksik veriler (belirsiz baz çağrıları) için izin verildi. Zaman içinde popülasyon büyüklüğündeki değişikliklerin tahmin edilmesine izin vermek ve Majander vd.'nin ardından26, on gruplu bir Bayes skyline birleşik dağılım modeli ağaç önseli olarak kullanıldı. Saat modeli seçiminin ardından, ortalama oran için 0 ile 1 ikame/site/yıl arasında düzgün önsele sahip ilişkisiz log-normal gevşemiş saat modeli kullanıldı103 (Ek Veriler, BEAST2_UCLN.xml). Diğer tüm parametreler varsayılan olarak ayarlandı (BEAUti v.2.6.7)38. Analiz, 300 milyon yinelemeyi içeren bir MCMC ile her 30.000 adımda örnekleyerek çalıştırıldı. Yinelemelerin %10'u yakma süresi olarak elendi ve Tracer v. 1.7.1104'te yakınsama değerlendirildi ve tüm etkili örneklem boyutu (ESS) değerlerinin 200'ün üzerinde olduğundan emin olundu. Saat oranı ve TMRCA tahminlerinin veriler tarafından yönlendirildiğinden emin olmak için aynı MCMC özelliklerini kullanarak parametrelerin önsel dağılımını da örneklendirdik (Ek Şekil 19). Treeannotator105 kullanılarak ağaçların posterior dağılımından maksimum klad güvenilirliği (MCC; Şekil 4) ağacı oluşturuldu. Zaman içinde bakteri popülasyon büyüklüğünün tahminlerini görselleştirmek için özel bir R komut dosyası kullanılarak örneklenen ağaçlar ve popülasyon parametreleri bir Bayes skyline grafiği (Genişletilmiş Veri Şekil 4) oluşturmak için kullanıldı.
Ek olarak, gözlemlenen evrimsel oranların dikkate alınan zaman ölçeğine bağlı olarak değişebileceği gerçeğini hesaba katmak için, BEAST 1.10.442,106'da uygulanan zaman bağımlı oran (TDR) modelini kullanarak ikinci bir analiz gerçekleştirdik (Ek Veriler, BEAST1_TDR.xml). Ancak, ikincisi, ilişkisiz gevşemiş saat modelinin yaptığı gibi soy hatları arası oran değişimini içeremez. 100 yıl önce ve 1000 yıl önce bir geçişe sahip üstel bir yapıya sahip üç dönemli bir model kullandık. Her dönemin orta noktası referans noktası olarak kullanıldı (son dönem için 5000 yıl önce alındı). Sırasıyla kesme ve eğim katsayıları için ortalama = −14,5 ve 0 ve s.d. = 5 ve 5 olan normal önselleri kullandık (sırasıyla zaman 0'da yaklaşık 5.10–7 ikame/site/yıl ortalamasına ve ilişkinin yönü hakkında önsel varsayıma karşılık gelir). Diğer model özellikleri yukarıda açıklanan ana analizle aynıydı (Ek Şekil 20). Tarihleme analizlerinin sonuçları Ek Tablo 29'da sunulmuştur.
Raporlama özeti